การประยุกต์ใช้ Nanopore Sequencing
การอ่านลำดับพันธุกรรมสายยาว ความสะดวกในการพกพา และความสามารถในการทำ direct RNA sequencing ของเครื่อง ONT นั้นช่วยในงานได้หลายด้าน (Fig. 5)
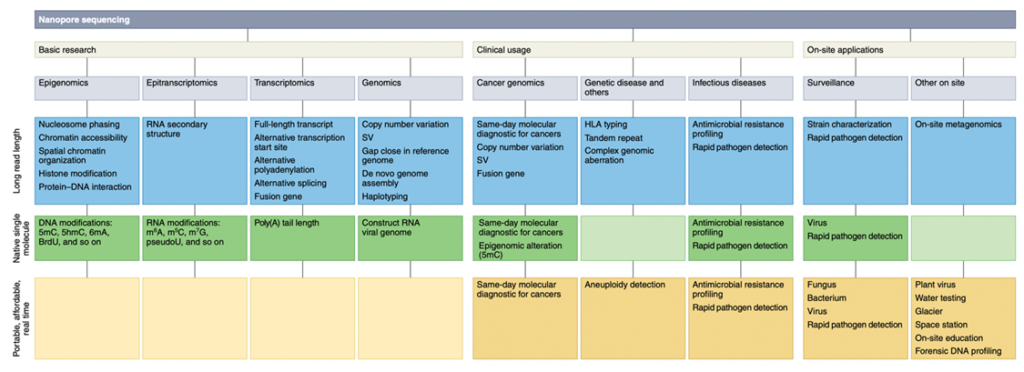
Fig. 5 | การประยุกต์ใช้งานของ ONT sequencing แบ่งเป็น 3 กลุ่มหลัก (งานวิจัยพื้นฐาน, การใช้งานทางคลินิก และ การประยุกต์ใช้งานนอกสถานที่) การแบ่งกลุ่มหลังจากนี้มักจะเป็นการแบ่งจากหัวข้อเฉพาะเจาะจง และวงการเฉพาะตามสัดส่วนของงานตีพิมพ์ บางการประยุกต์ใช้งานแบ่งออกไปอีกเป็นสองกลุ่มย่อย เช่น SV detection และ rapid pathogen detection การประยุกต์ใช้นั้นยังได้ถูกจัดโดยอาศัยจุดแข็งของ ONT sequencing เป็น 3 ชั้น (1) ความยาว (long read), (2) โมเลกุลต้นฉบับ (native single molecule) และ (3) การถือไปใช้ที่อื่นได้, ซื้อหาได้ และเห็นผลได้แบบทันที (real time) ความกว้างของแต่ละชั้นเชื่อมโยงกับจำนวนงานตีพิมพ์ บางการประยุกต์ใช้งานทั้ง 3 ชั้น (ตัวอย่างเช่นการทำโปรไฟล์ antimicrobial resistance) ใน ‘Fungus’ อย่างเช่น Candida auris ใน ‘bacterium’ อย่างเช่น Salmonella, Neisseria meningitidis และ Klebsiella pneumoniae และใน ‘virus’ อย่างเช่นไวรัสในกลุ่มที่ทำให้มีอาการทางระบบทางเดินหายใจเฉียบพลัน ได้แก่ coronavirus 2 (SARS-CoV-2), ebola, Zika, Venezuelan equine encephalitis, yellow fever, Lassa fever และ dengue: HLA, human leukocyte antigens
การเติมเต็มช่องว่างของจีโนมที่จะใช้อ้างอิง (Closing gaps in reference genomes) การประกอบข้อมูลจีโนมเป็นหนึ่งในการใช้งานหลักของเครื่อง ONT (ประมาณ 30% ของงานตีพิมพ์; Fig. 5) สำหรับสปีชี่ส์ที่มี reference genomes อยู่แล้ว เครื่อง ONT long reads มีประโยชน์ในการทำข้อมูลจีโนมให้สมบูรณ์ โดยเฉพาะจีโนมอ้างอิงมนุษย์ ONT read สามารถครอบคลุมช่องว่างจีโนมที่ขาดไปได้ถึง 12 ช่องว่าง (แต่ละช่องว่างมีขนาดใหญ่กว่า >50 kb) ใช้ในการอ่านความยาวของจำนวนซ้ำบนเทโลเมียร์ ( telomeric repeats) และสามารถประกอบข้อมูลในบริเวณ centromeric region ของโครโมโซม Y ในมนุษย์ได้อีกด้วย นอกจากนี้ ONT ยังสามารถทำให้ได้ข้อมูลสมบูรณ์ในการประกอบ telomere-to-telomere บนโครโมโซม X ของมนุษย์อีกด้วย รวมถึงการปรับโครงสร้าง (reconstruction) ของ centromeric satellite DNA array ขนาดประมาณ 2.8Mb และช่วยปิดช่องว่างทั้งหมดถึง 29 gaps (ทั้งหมด 1.1Mb) ที่มีในรายงานของ Telomere-to-Telomere Consortium ซึ่งได้จีโนมมนุษย์ที่สมบูรณ์ครั้งแรก (T2T-CHM13) เป็นจำนวนเบสทั้งหมด 3.055 Gb การศึกษาจีโนมของ Caenorhabditis elegans ที่มีปริมาณข้อมูลกว่า 2 ล้านเบส ได้พบข้อมูลที่ทำให้มีความถูกต้องมากขึ้น จากการระบุบริเวณ repetitive regions ได้มากขึ้นเมื่อใช้ ONT long reads และได้มีรายงานความคืบหน้าที่คล้ายกันในสิ่งมีชีวิตอื่นที่ใกล้เคียงกัน เช่น Escherichia coli, Saccharomyces cerevisiae, Arabidopsis thaliana และ Drosophila species จำนวน 15 สายพันธุ์ย่อย) เหมือนกันกับในสิ่งมีชีวิตกลุ่ม non-model organisms ที่ได้มีการค้นคว้าลักษณะเฉพาะของ tandem repeats ในจีโนมของข้าวสาลีสำหรับทำขนมปังและพัฒนาอย่างต่อเนื่องเพื่อให้ได้จีโนมที่สมบูรณ์ ของ Trypanosoma cruzi (ปรสิตที่เป็นสาเหตุของโรค Chagas)
การสร้างจีโนมอ้างอิงขึ้นมาใหม่ (Building new reference genomes) ONT long reads ใช้มากในการประกอบข้อมูลจีโนมที่ศึกษาใหม่ โดยเน้นหนักไปที่การทำจีโนมอ้างอิงในสิ่งมีชีวิตอื่นๆ กลุ่มที่ไม่ใช่สิ่งมีชีวิตตัวแทนอ้างอิง (non-model organisms) ตัวอย่างเช่น มีการนำข้อมูลที่ได้จาก ONT เดี่ยวๆมาเพื่อใช้หาจีโนมครั้งแรกของ Rhizoctonia solani (สปีชี่ส์ของเชื้อราก่อโรคชนิดหนึ่ง ที่เป็นสาเหตุของโรคในพืชทางการเกษตร) จากนั้นก็มีการนำไปทำ hybrid sequencing (ONT และ Illumina) เพื่อนำไปใช้หาข้อมูลจีโนมเบื้องต้นของ Maccullochella peelii (ปลาน้ำจืดที่ใหญ่ที่สุดของออสเตรเลีย) และ Amphiprion ocellaris (ปลาการ์ตูน) ในกรณีที่ซับซ้อนมากขึ้น ONT long reads ได้นำไปรวมกับเทคนิคใดเทคนิคหนึ่ง Illumina short reads, PacBio long reads, 10x Genomics linked reads, optical mapping (Bionano Genomics) และ spatial distance โดย Hi-C เพื่อที่จะรวบรวมข้อมูลให้ได้จีโนมอ้างอิงเป็นครั้งแรกในสปีชี่ส์ที่หลากหลาย เช่น Maniola jurtina (ผีเสื้อทุ่งหญ้าสีน้ำตาล เป็นตัวแทนของพันธุศาสตร์ระบบนิเวศ) Varanus komodoensis (ตัวเงินตัวทองขนาดใหญ่) Pavo cristatus (นกประจำชาติของอินเดีย) Panthera leo (สิงโต) และ Eumeta variegate (มอดหนอนไหม) นอกจากนี้ได้มีการใช้ ONT ในการทำการหาลำดับ RNA โดยตรง (direct RNA sequencing) ได้ถูกใช้ในการหาจีโนมของ RNA ไวรัส โดยที่ไม่ต้องผ่านขั้นตอนแปลงกลับให้เป็น DNA ก่อน (reverse transcription) ได้แก่ Mayaro virus, Venezuelan equine encephalitis virus, chikungunya virus, Zika virus, vesicular stomatitis Indiana virus, Oropouche virus, influenza A และ human coronavirus ส่วนในจีโนมไวรัสที่มีจีโนม DNA/RNA ขนาดเล็ก อาจไม่จำเป็นต้องใช้ข้อมูลความยาวระดับ long read เช่น จีโนม human coronavirus ที่พบว่ามีขนาดจีโนมประมาณ 27 กิโลเบส ในกรณีนี้ ที่มีการระบาดของ SARS-CoV-2 เทคโนโลยี ONT sequencing ได้นำมาใช้ในการปรับโครงสร้างลำดับเบสในจีโนมแบบเต็มสายของ SARS-CoV-2 ผ่านการหาลำดับเบสแบบ cDNA และ direct RNA เพื่อให้ได้ข้อมูลที่มีประโยชน์ต่องานทางด้านชีววิทยา การเปลี่ยนแปลงของสายพันธุ์ และความสามารถในการก่อโรคของไวรัส
ปัจจุบัน ปริมาณข้อมูลที่เพิ่มขึ้น ความยาวที่อ่านได้ และความถูกต้อง ของข้อมูล ONT ช่วยทำให้มีเวลามากขึ้น และต้นทุนต่ำลง ในการทำการรวบรวมข้อมูลจีโนม (genome assembly) ในทุกๆขนาดของจีโนม ตั้งแต่แบคทีเรียที่มีหลายล้านเบส (megabases) ส่วนพวกแมลงวันผลไม้ ปลา เลือดหอย กล้วย กะหล่ำปลี และวอลนัท จีโนมเหล่านี้มีหลายร้อยล้านเบส (hundreds of megabase) ยังมีพวก Komodo dragon, Steller sea lion ผักกาดหอม และ giant sequoia ที่มีจีโนมในระดับ พันล้านต้นๆ (gigabases) จนถึง coast redwood และดอกทิวลิป ที่มีจีโนมขนาดหลายๆพันล้านเบส (27–34Gb) ส่วนในจีโนมมนุษย์นั้น (3.055 พันล้านเบส) ใช้เครื่องรุ่น PromethION ร่วมกับ flow cells 3 อันเท่านั้น เพื่อใช้ในการหาลำดับเบส ใช้ระยะเวลาการวิเคราะห์ด้วยคอมพิวเตอร์น้อยกว่า 6 ชั่วโมง
แปลโดย : Champ Sarawut
ที่มา: Nature biotechnology review article (https://doi.org/10.1038/s41587-021-01108-x)

