กลยุทธ์เพิ่มเติมที่จะช่วยเพิ่มความถูกต้องให้แก่การหาลำดับเบส นอกเหนือจากการพัฒนาในส่วน nanopore และ motor protein แล้ว ยังมีกลยุทธ์อีกหลายอย่างที่ถูกพัฒนาขึ้นเพื่อเพิ่มความถูกต้องในการหาลำดับเบส คุณภาพของข้อมูลสามารถพัฒนาได้โดยการอ่าน dsDNA แต่ละสายซ้ำหลายๆรอบ เพื่อให้ได้ลำดับที่พบบ่อย (consensus sequence), คล้ายกับกลยุทธ์ที่ใช้กับ ‘circular consensus sequencing’ ที่ใช้วิธีหาลำดับแบบ single-molecule long-read sequencing อย่างเช่นยี่ห้อ Pacific Biosciences (PacBio)
เวอร์ชั่นแรกๆ ONT ใช้วิธีที่ชื่อว่า 2D library preparation ซึ่งมีการการหาลำดับ dsDNA สองรอบ โดย dsDNA ทั้ง 2 เส้น จะเชื่อมต่อเข้าด้วยกันด้วย hairpin adapter โดยมี motor protein ช่วยนำทางแล้วเริ่มที่เส้นแรก (template) จากนั้นจึงตามด้วย hairpin adapter แล้วไปต่อที่เส้นที่สอง (complement) (Fig. 3d, ซ้าย) หลังจากลบ hairpin sequence ออก ผลการอ่านทั้งสาย template และ complement จะเรียก 1D reads เมื่อนำไปทำเป็น consensus sequence จะเรียกมันว่า 2D read ที่มี accuracy สูงกว่า ตัวอย่างเช่นการใช้ flow cell รุ่น R9.4 ค่าเฉลี่ยของ accuracy เมื่อทำ 2D reads จะเท่ากับ 94% ซึ่งดีกว่า 86% เมื่อทำแบบ 1D reads (Fig. 2b).
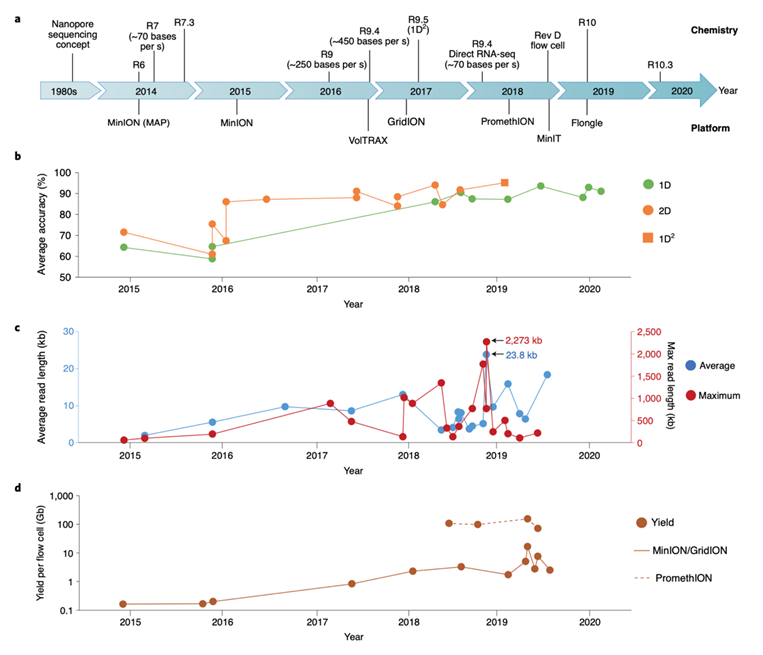
Fig. 2 | การพัฒนาข้อมูล ONT sequencing อย่างต่อเนื่อง (a), ลำดับเวลาของเทคนิคและรุ่นที่ออกมาจำหน่าย (b)
ค่า Accuracy ของ 1D, 2D และ 1D2 (c), ค่าเฉลี่ยและความยาวสูงสุดที่อ่านได้ ตัวอย่างเช่น ปลายปี 2019 ความยาวสูงสุดเฉลี่ยได้ถึง 23.8 กิโลเบส (kb) ซึ่งมีการใช้เทคนิค DNA extraction พิเศษ ซึ่งพบว่าสายที่ยาวที่สุดยาวถึง 2,273 kb
ความยาวที่อ่านได้เมื่อใช้1D reads (d), ปริมาณข้อมูลที่ได้ต่อ flow cell (log10 scale ในแกน y)
ในเดือนพฤษภาคม ค.ศ. 2017 ทาง ONT ได้เพิ่มเทคนิคที่เรียกว่า 1D2 ออกมาพร้อมกับ flow cell รุ่น R9.5 ในเทคนิคนี้ แทนที่จะใช้ hairpin adapter สาย DNA แต่ละเส้นได้ถูกติด special adapter แทน (Fig. 3d, ขวา) ซึ่ง adapter พิเศษนี้มีโอกาส 60% ที่จะถูกจับที่เส้นที่สอง (complement) อย่างต่อเนื่องหลังจากที่ทำการ sequence เส้นแรก (template) เสร็จ ทำให้ได้ consensus sequence คล้ายกับการทำ 2D ซึ่งค่าเฉลี่ยของ accuracy เมื่อใช้ 1D2 reads นั้นได้ถึง 95% (R9.5 nanopore) (Fig. 2b) ความต่างที่ไม่เหมือน 2D library ก็คือเส้น complement ใน 1D2 อาจจะไม่ได้ sequence ตามเส้น template ทั้งหมด ส่งผลให้สร้าง consensus sequence ได้ไม่สมบูรณ์นัก อย่างไรก็ตาม ONT ไม่ได้สนับสนุนการใช้งาน 2D and 1D2 ต่อหลังจากนั้น ปัจจุบันสำหรับ DNA sequencing แล้ว ONT สนับสนุนเฉพาะวิธีแบบ 1D ที่มีการติด adapter แยกระหว่าง dsDNA แต่ละเส้น (Fig. 3d, กลาง). แต่ในขณะเดียวกัน ได้มีการเพิ่ม accuracy โดยการพัฒนา base-calling algorithms เพิ่มเติม ตัวอย่างเช่นมีการใช้ flow cell รุ่น R7.3 ค่า accuracy ที่ได้จากการทำ 1D read ได้เพิ่มขึ้นจาก 65% ไปเป็น 70% เมื่อใช้ Nanocall และ 78% เมื่อใช้ DeepNano (Markov model (HMM))
เพิ่มความยาวที่สามารถอ่านได้ (Extending read length) ความยาวสูงสุดที่อ่านได้ในปี 2018 ได้ถึง 2.273megabases (Mb) ซึ่งความยาวที่อ่านได้ในการใช้ ONT ขึ้นอยู่กับขนาดโมเลกุลที่นำมาทำ sequencing library ซึ่งมีหลายวิธีที่จะทำได้ เพื่อให้ได้ high-molecular-weight (HMW) DNA เช่น spin columns (Genomic DNA Purification kit), gravity-flow columns (HMW DNA kit), magnetic beads (HMW DNA kit), phenol–chloroform, dialysis and plug extraction (Fig. 3a). HMW DNA สามารถลดขนาดให้เป็นสายสั้นตามที่ต้องการได้โดยใช้ sonication หรือ needle extrusion หรือ transposase cleavage (Fig. 3a) แต่อย่างไรก็ตามสายสั้นๆที่มากเกินไป จะทำให้ sequencing yield ลดลง เพราะจะเข้าไปรบกวนทั้งขั้นตอน adapter ligation และ translocation สายสั้นที่ไม่ต้องการจะถูก sequence ได้มากกว่าสายยาว ในการที่จะกำจัด DNA สายสั้นนั้น มีกลายวิธีที่ใช้ได้ เช่น gel-based BluePippin system จาก Sage Science หรือ magnetic beads และ the Short Read Eliminator kit จาก Circulomics ซึ่งเป็นที่นิยมมาใช้ในการคัดเลือกขนาดความยาว DNA ที่ต้องการ (Fig. 3a).
การหาลำดับเบส RNA (Sequencing RNA) เครื่องONT สามารถนำไปประยุกต์ใช้กับการทำ direct sequence native RNA ได้ วิธีการนี้ใช้ library preparation พิเศษที่ primer จะไปเชื่อมต่อด้าน 3′ end ของ native RNA ตามด้วย adapter ที่จะไปต่อตรง โดยไม่ต้องทำ conventional reverse transcription (Fig. 3c). หรืออีกทางเลือกหนึ่ง จะสังเคราะห์เป็น cDNA ก่อน เพื่อให้ได้ RNA–cDNA hybrid duplex แล้วจึงต่อด้วย adapter ทีหลัง
กลยุทธ์ที่ดีควรจะใช้ตัวอย่างที่ถูกรบกวนน้อยและใช้อย่างรวดเร็ว โดยเฉพาะตัวอย่างที่เพิ่งเก็บมา ถ้าสามารถดำเนินการได้ทันทีก็จะได้ library ที่เสถียร ปริมาณข้อมูลที่ดี และสายที่ยาวมากขึ้น ถ้าจะให้ดีควรมีเพียงสาย RNA ที่จะผ่านเข้าไปใน nanopore และอีกอย่างหนึ่งก็คือ การทำ direct sequencing กับ RNA ไม่สามารถสร้าง consensus sequence ได้ (แบบที่ใช้วิธี 2D หรือ 1D2) ซึ่งต่างจาก DNA sequencing ทำให้โดยปกติแล้ว direct RNA sequencing จะได้ accuracy เฉลี่ย 83–86%
สิ่งที่เหมือนกับ conventional RNA sequencing อื่นๆ คือ ONT สามารถทำ cDNA sequencing โดยใช้ cDNA synthesis (เช่น TeloPrime Full-Length cDNA Amplification kit of Lexogen) แล้วตามด้วยการเพิ่มจำนวนด้วย PCR (Fig. 3b) นอกจากนี้ ONT เสนอวิธีทำ direct cDNA sequencing โดยไม่ต้องทำ PCR อีกด้วย ซึ่งต่างจากการทำ cDNA sequencing โดยทั่วไป เนื่องจากจะช่วยลด PCR amplification bias แต่ก็จำเป็นต้องใช้ปริมาณตัวอย่างเริ่มต้นจำนวนมากและใช้เวลาเตรียม library นานเช่นกัน จึงไม่เหมาะกับงานทางด้านคลินิก แต่ก็มีงานวิจัยบางส่วนที่ออกมาบอกว่าการใช้ ONT มาหาลำดับ RNA, cDNA หรือ PCR-cDNA นั้น สามารถบ่งชี้และหาปริมาณ gene isoforms ได้
การเพิ่มจำนวนตัวอย่าง (Increasing throughput) นอกจาการเพิ่มความยาวการอ่านลำดับเบสและความถูกต้องแล้ว การเพิ่มจำนวนตัวอย่างก็เป็นอีกหนึ่งความสำคัญ ที่ ONT นำมาพิจารณา เพื่อตอบสนองความจำเป็นต่องานแต่ละขนาด ทาง ONT ได้ปล่อย platforms มาหลายแบบ (MinION, GridION, PromethION, Flongle และอื่นๆ) ข้อมูลที่คาดหวังจะได้ออกมาขึ้นอยู่กับ (1) จำนวน active nanopores, (2) ความเร็ว DNA/RNA translocation เข้าไปใน nanopore และ (3) ระยะเวลาการทำงาน (running time)
ในช่วงต้นผู้ใช้งาน MinION แจ้งผลมาว่าได้ปริมาณข้อมูลประมาณล้านเบส จำนวนหลักร้อยเส้น ต่อ flow cell แต่ปัจจุบันได้ถึง ~10–15 กิกะเบส (Gb) (Fig. 2d, เส้นทึบ) สำหรับ DNA sequencing ที่ใช้สารเคมีตัวใหม่ที่เร็วกว่า (เพิ่มจาก ~30 เบสต่อวินาที เมื่อใช้ R6 ไปเป็น ~450 เบสต่อวินาที โดยใช้ R9.4) และทำงานได้นานขึ้นด้วยโดยใช้ชิป ASIC เครื่องรุ่นอื่น เช่น PromethION ทำงานกับ flow cells ได้มากกว่า มี nanopores ต่อ flow cell มากกว่า โดยมีรายงานว่าได้ถึง 153 Gb จาก PromethION flow cell อันเดียว มีอัตราความเร็วประมาณ ~430 เบสต่อวินาที (Fig. 2d, เส้นประ) ส่วนในกรณีทำ direct RNA sequencing ปัจจุบัน พบว่าอ่านได้ประมาณ 1,000,000 reads (1–3 Gb) ต่อ MinION flow cell เนื่องจากว่าความเร็วในการค่อนข้างช้ากว่ามาก (~70 เบสต่อวินาที)
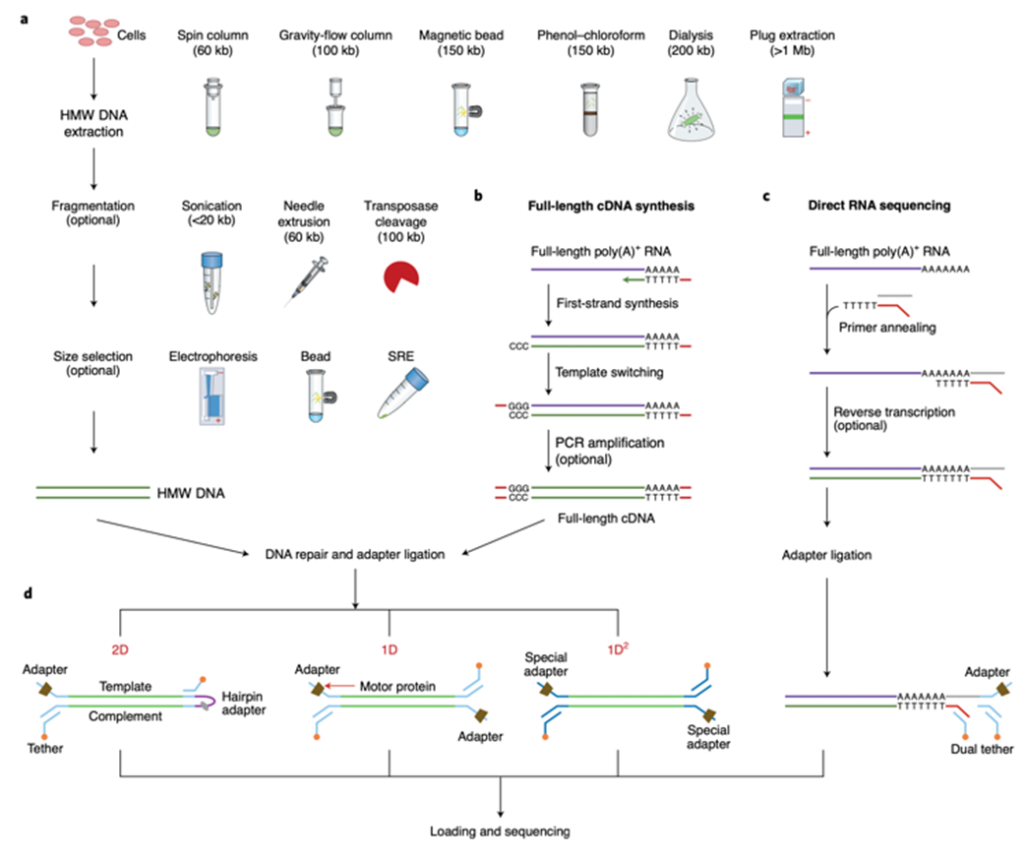
Fig. 3 | ขั้นตอนการทำ library preparation ของ ONT sequencing a. เทคนิคพิเศษที่ใช้เพื่อให้ได้ ultralong genomic DNA sequencing, รวมถึง HMW DNA extraction, fragmentation และ size selection b. การสังเคราะห์ Full-length cDNA เพื่อการทำ direct cDNA sequencing (ไม่ทำขั้นตอน PCR amplification) และ PCR-cDNA sequencing (ทำขั้นตอน PCR amplification) c. การเตรียม Direct RNA-sequencing library โดยใช้และไม่ใช้ขั้นตอน reverse transcription step, ซึ่งมีเพียง RNA strand ที่จะต่อเข้ากับ adapter และ RNA strand เท่านั้นที่จะถูก sequence d. กลยุทธ์ที่แตกต่างกันในการทำ library สำหรับ DNA/cDNA sequencing ได้แก่ 2D, 1D และ 1D2 : ใช้ SRE – short read eliminator kit (Circulomics)
แปลโดย : Champ Sarawut
ที่มา: Nature biotechnology review article (https://doi.org/10.1038/s41587-021-01108-x)

