การออกแบบของ ONT มีแนวคิดตั้งแต่ปี ค.ศ. 1980 ได้ค้นพบโปรตีนชนิดใหม่ที่มีลักษณะเป็นช่องเล็กๆระดับนาโน (nanopore) และโปรตีนที่มีความสามารถในการขับเคลื่อน nucleotide (motor protein) โปรตีนที่มีลักษณะเป็นช่องนั้นชื่อว่า α-Hemolysin ซึ่งเป็นโปรตีนบนเยื่อหุ้มเซลล์ของเชื้อ Staphylococcus aureus มีขนาดเส้นผ่านศูนย์กลางประมาณ ~1.4nm ถึง ~2.4nm เป็นนาโนพอร์ตัวแรกที่พบว่ามีความสามารถในการตรวจวัดและจดจำประจุไอออนที่เกิดจาก RNA และ DNA ประเภท homopolymers ได้ แต่เนื่องจากเราต้องการจำแนกชนิดเบสของ DNA ทั้ง 4 ตัว (A, T, C, G) จึงต้องทำการดัดแปลงโปรตีน α-hemolysin ดั้งเดิม ให้มีความสามารถเพิ่มเติมในส่วนนี้ ซึ่งก็มีการค้นพบที่คล้ายกันนี้ จากเชื้อ Mycobacterium smegmatis porin A (MspA) ที่มีขนาดเส้นผ่านศูนย์กลาง ใกล้เคียงกัน ที่ประมาณ 1.2 nm กุญแจสำคัญคือการทำให้อัตราสัญญาณต่อสิ่งรบกวน (signal-to-noise) ดีขึ้น จะต้องทำให้ DNA เคลื่อนที่ผ่าน nanopore ช้าลงและควบคุมได้ จึงได้มีการทดลองใช้เอนไซม์ phi29 DNA polymerase หลังจากทดลองพบว่า phi29 DNA polymerase มีประสิทธิภาพในการควบคุมการไหลผ่านของดีเอ็นเอเข้าไปใน nanopore ได้อย่างมีประสิทธิภาพ และได้เรียกเอนไซม์ตัวนี้ว่า motor protein ซึ่งอันที่จริงแล้ว motor protein ตัวนี้ เป็นส่วนที่ทำให้การประยุกต์ใช้สมบูรณ์ เพราะว่าในเดือนกุมภาพันธ์ปี 2012 มีการทดลองแบ่งเป็นสองกลุ่ม (กลุ่มที่ใช้และไม่ใช้ phi29 เป็น motor protein เชื่อมต่อเข้ากับ α-hemolysin และ MspA) เมื่อทำการบันทึกสัญญาณไอโอนิค ของโมเลกุล DNA สายเดี่ยว พบว่า motor protein ช่วยลดความผันผวนในกระบวนการเคลื่อนที่ของสาย DNA และส่งผลให้ข้อมูลที่ได้มีคุณภาพดีขึ้น จากนั้นในเดือนเดียวกันในปี 2014 ทาง ONT ก็ได้ทำการเปิดตัว nanopore sequencing เครื่องแรก คือ MinION และได้จำหน่ายอย่างเป็นทางการในปี 2015 และนอกจาก ONT แล้ว ก็ยังมีบริษัทที่ใช้เทคโนโลยีโปรตีนนาโนพอร์นี้อีกเช่นกัน เช่น Genia Technologies (Nano-SBS), NobleGen Biosciences (optipore system) และ Quantum Biosystems (SBET)
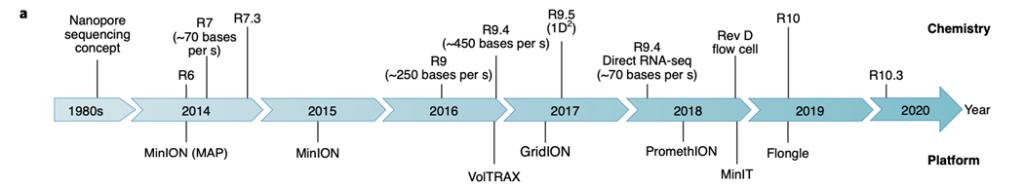
หลังจากนั้น ONT ได้มีการพัฒนา nanopore และ motor protein อย่างต่อเนื่อง และได้ นำเสนอออกมา 8 เวอร์ชั่น ได้แก่ R6 (มิ.ย. 2014), R7 (ก.ค. 2014), R7.3 (ต.ค. 2014), R9 (พ.ค. 2016), R9.4 (ต.ค. 2016), R9.5 (พ.ค. 2017), R10 (มี.ค. 2019) และ R10.3 (ม.ค. 2020) (Fig. 2a) โปรตีนทั้งตัวดั้งเดิมและตัวที่ผ่านการดัดแปลงในเวอร์ชั่น R6, R7, R7.3, R10 และ R10.3 นั้นทาง ONT ไม่มีการเปิดเผยข้อมูลเพิ่มเติม แต่ในส่วนของ R9 พบว่าประสบความสำเร็จในแง่ของความเร็วในการ sequence และความถูกต้อง (accuracy) ที่มากขึ้น (~87%) โดยเวอร์ชั่นนี้ได้มีการการเพิ่ม Curlin sigma S-dependent growth subunit G (CsgG) จาก Escherichia coli ทำให้ nanopore เวอร์ชั่นนี้มีความเร็วในการเคลื่อนที่ประมาณ 250 เบสต่อวินาที เวอร์ชั่นต่อมาจึงได้ใช้ CsgG และ motor protein ชนิดใหม่ ในเวอร์ชั่น R9.4 ที่ได้ความถูกต้องมากขึ้น (accuracy ~85-94%) และมีความเร็วการอ่านมากขึ้น (~450 เบสต่อวินาที) และถัดมา R9.5 ได้ถูกนำเสนอเป็นลำดับถัดมา ด้วยกลยุทธ์ใหม่ที่ชื่อว่า 1D2 sequencing ที่จะมีการวัด DNA สายเดี่ยวสองครั้ง
แต่อย่างไรก็ตาม เวอร์ชั่น R9.4 และ R9.5 ยังคงมีความยากในการอ่านลำดับเบสที่ซ้ำกันยาวมากๆ (very long homopolymer) เนื่องจากสัญญานไฟฟ้าที่รับจาก CsgG สามารถตรวจวัดเบสที่ซ้ำกันต่อเนื่องได้ประมาณ 5 homopolymer เท่านั้น จึงได้มีการพัฒนาเวอร์ชั่น R10 และ R10.3 จะมีบริเวณตรวจจับสองตำแหน่ง โดยมีจุดประสงค์เพื่อให้ได้ความถูกต้องในการอ่าน homopolymer มากขึ้น แต่ยังคงต้องมีการศึกษาเพิ่มเติมต่อไปเพื่อยืนยันแนวคิดนี้
แปลโดย : Champ Sarawut
ที่มา: Nature biotechnology review article (https://doi.org/10.1038/s41587-021-01108-x)

