เทคโนโลยี nanopore เป็นเทคโนโลยีการหาลำดับ DNA และ RNA แบบสายยาวต่อเนื่อง ปัจจุบันได้มีการพัฒนาความถูกต้อง (accuracy) ความยาว (length) และปริมาณ (throughput) โดย nanopore sequencing สามารถนำไปศึกษา genome, transcriptome, epigenome และ epitranscriptome นอกจากนี้ยังได้ถูกนำไปประยุกต์ใช้ในงาน genome assembly, full-length transcript detection, base modification detection และอื่นๆ ได้ อีกด้วย
Oxford Nanopore Technologies (ONT) เป็นเทคโนโลยีที่ใช้โปรตีนที่มีลักษณะเป็นช่องในระดับนาโน (nanopore) ซึ่งมีคุณสมบัติเป็น biosensor โดย nanopore จะถูกนำไปติดอยู่บนแผ่นโพลีเมอร์ที่มีความต้านต่อกระแสไฟฟ้า และใช้สารละลาย electrolyte จากนั้นควบคุมแรงดันไฟฟ้าให้คงที่เพื่อที่จะสร้างกระแสไอออนให้ไหลผ่าน nanopore จากฝั่งประจุลบ ‘cis’ ไปยังฝั่งประจุบวก ‘trans’ ซึ่งความเร็วในการเคลื่อนที่ของ nucleic acid ที่ไหลผ่านจะขึ้นอยู่กับ motor protein
ในส่วนของ motor protein จะมี helicase activity ช่วยในการแยกสาย double-stranded DNA หรือ RNA-DNA duplexes ให้กลายเป็น single-stranded ที่สามารถไหลผ่านเข้าไปใน nanopore ได้ ซึ่งการเปลี่ยนแปลงของกระแสไอออนขณะโมเลกุลไหลผ่านตรงบริเวณที่ตรวจจับจะถูกประมวลผลด้วย computational algorithm แล้วแสดงผลออกมาเป็น nucleotide sequence ทำให้ได้ real-time sequencing ของ single molecule
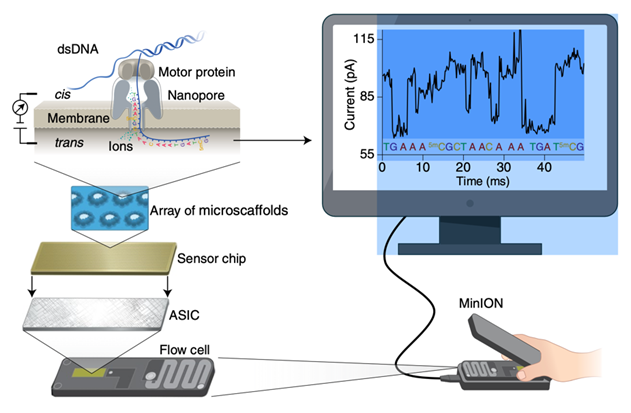
หลักการของ nanopore sequencing โฟลว์เซลล์ (minION flow cell) จะมีแบ่งพื้นที่เป็น 512 ช่อง แต่ละช่องมี 4 nanopores ดังนั้นจะมี nanopores ทั้งหมดจำนวน 2,048 ตัว ที่สามารถใช้หาลำดับ DNA หรือ RNA ตัว nanopore นี้ถูกนำไปติดไว้ใน polymer membrane ที่มีความต้านทานไฟฟ้า ซึ่งเชื่อมต่อกับ sensor chip ที่แต่ละช่องมีอิเล็คโทรดแยกกันและวัดกระแสแยกกันด้วยอุปกรณ์ที่ชื่อว่า application-specific integration circuit (ASIC)
กระแสไอออนจะมีการไหลผ่าน nanopore อย่างต่อเนื่อง เนื่องจากมีการใส่ volt อย่างคงที่ระหว่าง membrane โดยมีการกำหนดให้ฝั่ง trans นั้นมีประจุบวก ส่วนอีกฝั่ง cis จะมี motor protein ที่ทำให้ double-stranded DNA (dsDNA) (หรือ RNA–DNA hybrid duplex) ถูกแยกสายออกจากกัน เกิดเป็น single-stranded DNA หรือ RNA ที่มีประจุลบ ทำให้สามารถเริ่มไหลผ่านเข้าไปใน nanopore ได้
เมื่อ nucleotides ผ่านเข้าไปใน nanopore จะเกิดรูปแบบกระแสที่วัดได้เป็นกราฟ แล้วจะถูกนำไปแปลงผลให้เป็น nucleotide แต่ละตัว โดยมีความเร็วในการอ่านประมาณ 450 เบสต่อวินาที (สำหรับ R9.4 nanopore)
แปลโดย : Champ Sarawut
ที่มา: Nature biotechnology review article (https://doi.org/10.1038/s41587-021-01108-x)

