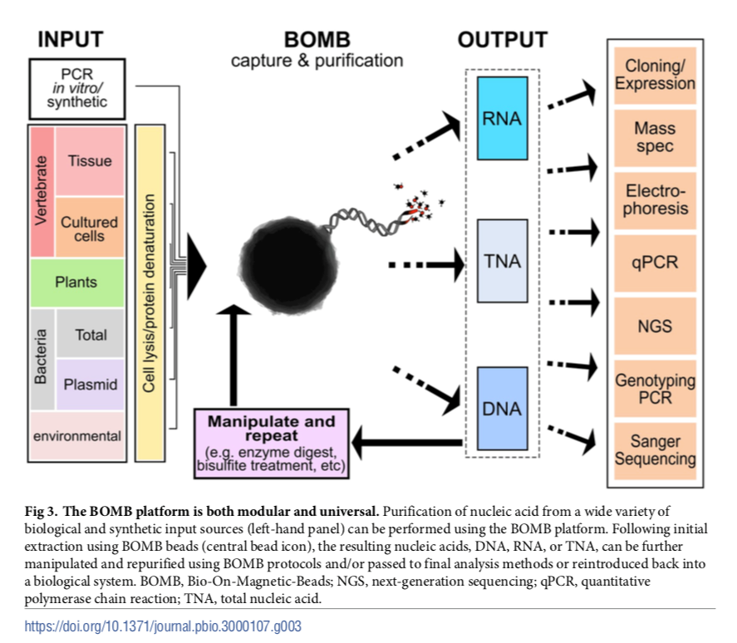
Simple synthesis of functionalised magnetic beads for nucleic acid manipulation
Protocol #1: Synthesis of ferrite core magnetic particles
การสังเคราะห์ Ferrite nanoparticles เพื่อใช้เป็นแกนหลักของเม็ด beads นั้นสามารถทำได้หลายวิธีตามการรายงานที่มีมาก่อนหน้า แต่ในครั้งนี้ เพื่อให้เป็นการง่ายและมีประสิทธิภาพ สารละลาย alkali จะถูกนำมาผ่านความร้อนเพื่อไล่ oxygen ออกก่อนนำมาใช้ จากนั้นให้นำสารละลายที่มีส่วนผสมของ FeCl3 และ FeCl2 (1:2 molar) มาหยดใส่ในสารละลาย alkali ที่เตรียมไว้ อย่างช้าๆ จนเกิดเป็น Ferrite paticles (Fe3O4) ซึ่งมีลักษณะเป็นตะกอนสีดำ หลังจากนั้น Ferrite particles ที่มีขนาดเส้นผ่านศูนย์กลางประมาณ 5-20 นาโนเมตร จะถูกนำไปล้างด้วย น้ำ DI เพื่อใช้ในการทดลองต่อไป
หมายเหตุ: ในกรณีที่ต้องการป้องกันการเกิดปฏิกิริยา oxidation ต่อ particles ควรนำ particles ที่ได้ไปเคลือบด้วย silica หรือ carboxyl ในทันทีหลังจากการถูกสังเคราะห์ หรือจะนำไปเก็บใน sodium oleate, polyvinylpyrrolidone (PVP) หรือสารเคมีอื่นๆดังที่มีการรายงานก่อนหน้าก็ได้ (S1 Appendix, BOMB protocol 1.1)
Protocol #2: Coating MNPs with silica
การเคลือบ particles ด้วย silica นั้นจำเป็นจะต้องใช้ขวดแก้วเป็นภาชนะสำหรับการเกิดปฏิกิริยา ด้วยเหตุผลหลักๆคือเป็นการป้องกันการเกิด oxidation ของ MNPs โดยเทคนิคที่ใช้ในการเคลือบ particles ในครั้งนี้คือ modified Stober method เป็นเทคนิคที่ทำการใส่ Tetraethyl orthosilicate (TEOS) ลงไปในสารละลาย เพื่อที่จะได้ถูก hydrolyse กลายเป็นชั้นของ SiO2 แล้วไปเคลือบลงบน เม็ด magnetic core หรือ Ferrite particles ที่เราสังเคราะห์ได้ ซึ่งความหนาของชั้น SiO2 ที่เคลือบลงบน particles นั้น จะขึ้นอยู่กับปริมาณของ TEOS ที่ใส่เข้าไปในปฏิกิริยา โดยที่ความหนาของชั้น SiO2 ที่เหมาะสมสำหรับการจัดการและทำให้ nucleic acid มีความบริสุทธิ์นั้นจะมีค่าอยู่ที่ประมาณ 400 นาโนเมตร (S1 Appendix, BOMB protocol 2.1)
Protocol #3: Coating MNPs with methacrylic acid (carboxyl coating)
นอกจากการนำ silica มาเคลือบลงบน particles แล้วนั้น การเคลือบด้วย methacrylic acid ก็ยังเป็นอีกวิธีหนึ่ง ที่สามารถคงความเสถียรของ MNPs ได้ ซึ่งขั้นตอนในการสังเคราะห์นั้นเริ่มจากการใส่ MNPs ลงไปใน Sodium dodecyl sulfate (SDS) แล้วเติม Polymethacrylic acid (PMAA) ตามลงไป เพื่อให้เกิดการฝังตัวลงบนพื้นผิวของ particles ด้วยปฏิกิริยา Free-radical retrograde precipitation polymerization (FRRPP) ทำให้ MNPs มีประจุเป็นลบอ่อนๆที่เกิดจากหมู่ carboxyl (S1 Appendix, BOMB protocol 3.1)
Immobilization racks for magnetic separation
การใช้ MNPs ในงานวิจัยนั้นมีประโยชน์และทำให้การทดลองมีความสะดวกขึ้นเป็นอย่างมาก เนื่องจากเราสามารถควบคุมให้ particles นั้นอยู่กับที่หรือจะให้เคลื่อนที่ไปในทิศทางใดก็ได้ ขึ้นอยู่กับทิศทางของสนามแม่เหล็กที่เราสร้างขึ้นมา ซึ่งวิธีที่ง่ายมากๆที่จะตรึง MNPs ให้อยู่กับที่และเหมาะสมในการทดลองที่สุดก็คือ การติดแถบแม่เหล็กลงบน racks สำหรับ microtube หรือ microplate แต่ว่าราคาของ racks ชนิดนี้ในท้องตลาดนั้นก็มีราคาสูงมาก อย่างไรก็ตามการทำ racks ชนิดนี้ขึ้นมาใช้เองก็สามารถทำได้โดยง่าย โดยที่วัสดุที่จำเป็นที่สุดก็คือ magnet นั่นเอง เพื่อที่จะทำให้สามารถแยก MNPs ออกจากสารละลายได้ไว high-quality neodymium magnet เกรด N42 หรือสูงกว่านั้นได้ถูกนำมาใช้ในการสร้าง racks ที่มีลักษณะเหมาะสมกับหลอดทดลองแบบต่างๆ จากวัสดุเหลือใช้, 3D-printed หรือ การใช้ laser-cut (Fig 1) ยกตัวอย่างเช่น เมื่อต้องการใช้ 8 strip PCR tubes หรือ 96-well plates เราก็สามารถนำแผ่นแม่เหล็กไปติดไว้บนกล่อง pipette tip ได้ สำหรับผู้ที่สนใจจะดูตัวอย่าง racks ก็สามารถเข้าไปดูได้ที่ (S1 Appendix, BOMB protocol A.1 และ A.2, BOMB files 1-4)
Protocol #4: Clean-up and size exclusion
โดยทั่วไปในการแยกและการทำให้ nucleic acid บริสุทธินั้น จำเป็นจะต้องใช้เอนไซม์ในการทำปฏิกิริยา อีกทั้งยังต้องเสียเวลาในขั้นตอนของการตกตะกอนด้วยเกลือและ alcohol ทำให้มีโอกาสที่จะสูญเสียดีเอ็นเอขนาดเล็กไปในระหว่างการทดลองได้ ดังนั้นการนำวิธี Silica column หรือ SPRI บน Magnetic beads มาใช้ จึงเป็นการช่วยให้ nucleic acid นั้นมีความบริสุทธิมากขึ้น เนื่องจากเป็นปฏิกิริยาที่เกิดขึ้นในหลอดทดลองเดียว และสามารถควบคุม binding condition ระหว่างเม็ด beads กับขนาดของ nucleic acid fragments ได้ ทำให้สามารถนำ nucleic acid ที่ได้มานั้นไปใช้ต่อในงานต่างๆได้โดยง่าย เช่น การทำ DNA library สำหรับ Next-generation sequencing
การเลือกขนาดของ fragments นั้นสามารถทำได้โดยการใช้ BOMB beads ที่เคลือบด้วย silica หรือ carboxyl ก็ได้ (S1 Appendix, BOMB protocol 4.1 and 4.2) แต่ว่า binding condition นั้นก็จะมีความแตกต่างกันออกไป โดยหลักการแล้ว DNA นั้นจะสามารถจับกับ carboxyl beads โดยการรวมตัวเป็นก้อน (crowding) ภายใต้ความเข้นข้นของ PEG-8000 และ NaCl ที่สูง ในขณะที่ DNA จะจับกับ silica beads โดยใช้ประจุลบของ Phosphate ผ่านการเหนี่ยวนำของ chaotropic salts ซึ่งการทดลองส่วนใหญ่นั้นจะใช้ guanidinium hydrochloride ในการจับกับ silica beads
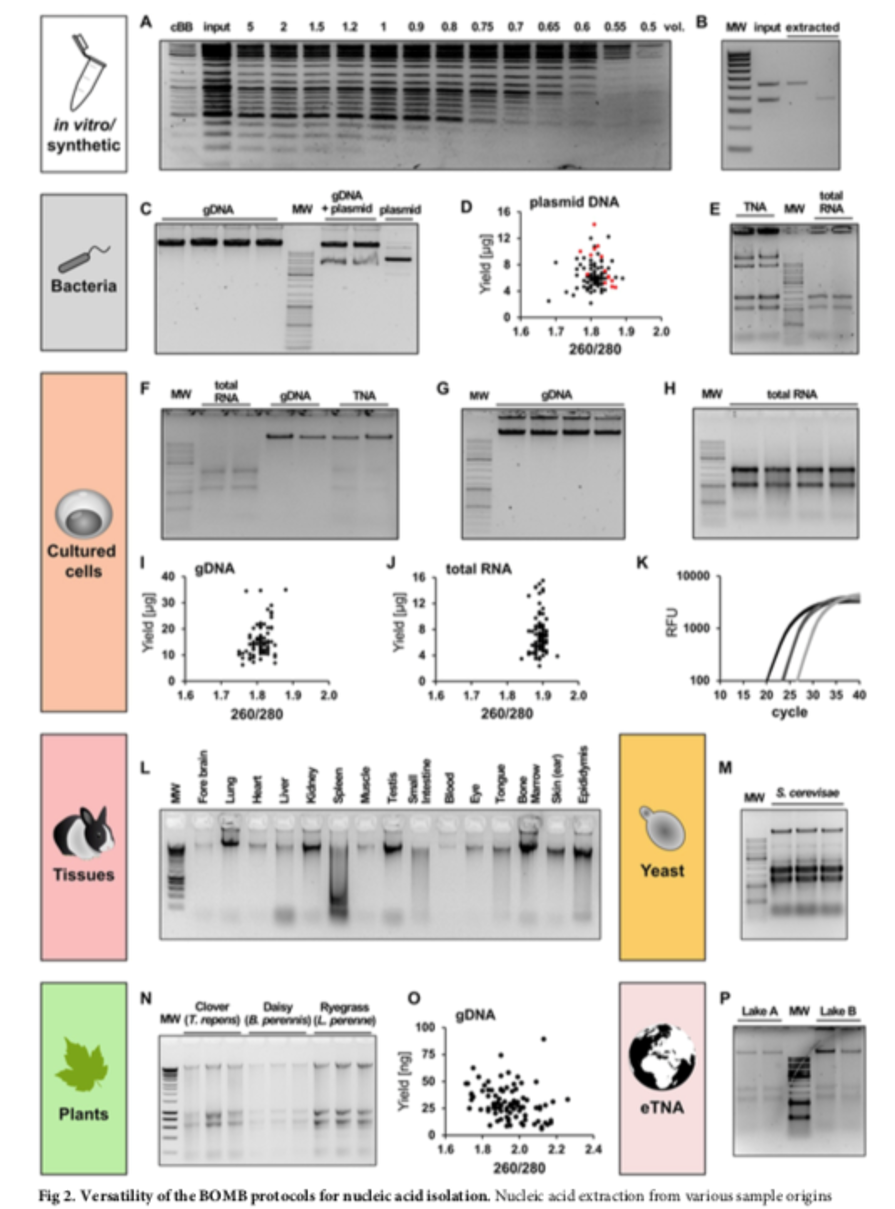
จากผลการทดลองโดยใช้ BOMB beads นั้นจะทำให้เราสามารถแยก DNA fragments ได้ตั้งแต่ 10 bp – 3,000 bp (Fig 2A and S2 Fig) ขึ้นอยู่กับปริมาณของ binding buffer ที่ใช้ ในระหว่างการสกัด DNA นอกจากนี้การที่สามารถใช้ 96-well plate ควบคู่ไปกับ magnetic rack ก็ยังเป็นการลดระยะเวลาในการทดลองได้ โดยที่ตัวอย่างประมาณ 200 ตัวอย่างนั้นถูกทำให้เสร็จด้วยตัวคนเดียวภายในเวลา 45 นาที ท้ายที่สุดวิธีการทำให้ nucleic acid บริสุทธิ์จาก agarose gels โดยใช้ BOMB ก็ได้ถูกพัฒนาขึ้นตาม Fig 2B, S1 Appendix, BOMB protocol 4.3
Protocol #5: Bacterial plasmid extraction
การสกัด plasmid จากแบคทีเรีย E.coli นั้นน่าจะเป็นหนึ่งในขั้นตอนพื้นฐานที่ใช้ในห้องปฏิบัติการทั่วไป ในช่วงปี ค.ศ.1960 วิธีการสกัด plasmid จากแบคทีเรีย ได้ถูกเผยแพร่เป็นครั้งแรกจากการใช้ alkaline lysis และได้ถูกพัฒนาต่อเรื่อยมาจนถึงปัจจุบัน โดยที่หลายๆบริษัทก็ได้นำหลักการของ silica-packed column มาใช้ในการแยก DNA ซึ่งจัดว่าเป็นเทคนิคที่มีประสิทธิภาพและน่าเชื่อถือ รวมถึงมีราคาที่สมเหตุสมผลอยู่ที่ประมาณ 1.5 € ต่อตัวอย่าง ทั้งนี้ เทคนิค silica-packed column ก็ยังไม่เหมาะสำหรับการแยก DNA ที่มีจำนวนตัวอย่างครั้งละมากๆ ทำให้ silica-coated magnetic beads ได้ถูกพัฒนาขึ้นมาเพื่อให้สามารถแยก DNA ในรูปแบบ high-throughput ได้ สำหรับการทดลองในครั้งนี้ แบคทีเรียจะถูกเพาะเลี้ยงใน 2.2 mL 96 well plate (S3A Fig) และนำมาสกัด DNA ด้วยเทคนิค modified alkaline lysis โดยที่ plasmid DNA จะถูกจับอยู่บน silica beads ส่วนองค์ประกอบของเซลล์อื่นๆก็จะถูกล้างออก ซึ่งผลการทดลองจากการสกัด plasmid (pUC19) ด้วยวิธีนี้ จะทำให้ได้ปริมาณของ DNA อยู่ที่ประมาณ 6 μg ต่อตัวอย่าง (Fig 2C and 2D) (S1 Appendix, BOMB protocols 5.1 ใช้ centrifuge ในการแยกเซลล์กับ plasmid และ 5.2 เป็นการใช้ silica-coted MNPs) หลังจากนั้นปริมาณของ plasmid DNA รวมไปถึงความบริสุทธิ์และคุณภาพที่ได้จากการสกัดด้วย BOMB นั้นจะถูกนำไปเปรียบเทียบกับวิธีอื่นๆ เพื่อศึกษาความเหมาะสมที่จะนำ DNA ที่ได้ไปใช้ใน restriction digestion, Sanger sequencing และการทดลองอื่นต่อไป จากการทดลองในครั้งนี้ทำให้ทราบว่าการใช้ silica-coated BOMB beads นั้น ช่วยให้สามารถสกัด plasmid DNA ได้ถึง 200 ตัวอย่าง ภายในเวลาประมาณ 3-4 ชั่วโมง ซึ่งเมื่อคำนวนเป็นค่าใช้จ่ายแล้วนั้นจะตกอยู่ที่ประมาณ 0.14 € ต่อตัวอย่าง (Table 1)
Cr. ละปิง
ติดตามได้ต่อ Ep.3
ผลิตภัณฑ์ที่เกี่ยวข้อง
MagTec ViroNA, Nano Magnetic beads for DNA/RNA Extraction, BIOENTIST, THAILAND

